¶ Biovia Materials Studio
Materials Studio es un software para simular y modelar materiales. Está desarrollado y distribuido por BIOVIA, una firma especializada en software de investigación para química computacional, bioinformática, química, simulación de dinámica molecular y mecánica cuántica.
Entorno de modelado y simulación en el área de materiales desarrollado por la compañía Dassault. Combina métodos tales como Mecánica Molecular (MM), Dinámica Molecular (DM), Mecánica Cuántica (QM) y modelado en mesoescala en un entorno fácil de usar. Puede ejecutarse en el marco de una arquitectura cliente/servidor, lo que permite no sólo ampliar el espectro de accesibilidad de usuarios, sino también mejorar el rendimiento y eficiencia de los procesos.
Los módulos disponibles son: Adsorption Locator, Amorphous Cell, Blends, Castep, Compass, Conformers, DFTB+, Discover, Dmol3, DPD, Equilibra, ForcitePlus, GULP, Kinetix, Mesodyn, Morphology, NMR Castep, ONETEP, Polymorph, QMERA, QSAR PLUS, Reflex, Sorption, Synthia y VAMP.
Para más información haz clic aquí
¶ Disponibilidad
- Máquinas personales
- Sistemas Operativos: Windows, Linux
- Conexión VPN Forticlient del CSIC
- Drago: 22.1 (MaterialsStudio/22.1.MaterialsStudio) (Sin pasarela)
- Rama: 0.2
- Drago: 24.1 (MaterialsStudio/24.1.MaterialsStudio)
- Rama: 0.3
- Tipo de software: restricted
- Categoría: chem
- Nota: Antes de usar este software debe pedirse el alta a calculo cientifico
¶ Licencia
Materials Studio puede usarse en Drago con licencia.
Licencia de tipo en red con licencias concurrentes.
¶ Uso
Materials Studio es una aplicación que funciona bajo la aproximación de cliente/servidor. El cliente es una interfaz gráfica que se ejecuta en Windows. El servidor es la parte que realiza el cálculos y puede estar instalado de forma local o en remoto. En el caso del cluster Drago solamente se puede ejecutar en modo cliente por lo que es necesario configurar una pasarela (gateway) a través del cliente.
Para más información sobre el uso y los métodos implementados en Materials Studio se recomienda consultar el manual (Help/Help Topic).
¶ Instalación en windows.
¶ Configuración de la pasarela.
¶ Ejemplos
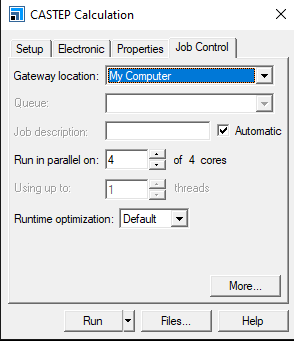
¶ Pasarela

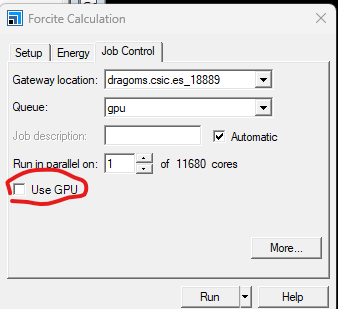
Solo se debe utilizar la cola GPU con los módulos Forcite y Mesocite. Marcar el check [Use GPU]
¶ Standalone
- Plantilla para un script
Como se puede ver en el video de ejemplo, se necesita la preparación de un script para ejecutar el trabajo.
Se asume que el usuario está familiarizado con el uso básico de slurm. Un tutorial esta disponible en la sección correspondiente
- Plantilla para un script en un nodo.
#!/bin/bash
#SBATCH -p generic
#SBATCH -N 1
#SBATCH --ntasks-per-node=<Número de procesadores a usar en el nodo, por ejemplo 16>
#SBATCH --job-name=<Nombre del trabajo, por ejemplo 01-Job>
#SBATCH --mem-per-cpu=<Cantidad de memoria asignada por cpu, por defecto 4g>
#SBATCH --constraint=MS
#Modulos necesarios para Materials Studio
module purge
module load intel/2020b
#Configuracion estandar, se recomienda no modificar
scontrol show hostname $SLURM_NODELIST
cd $SLURM_SUBMIT_DIR
export LC_CTYPE=en_US.UTF-8
export LC_ALL=en_US.UTF-8
echo $SLURM_SUBMIT_DIR
NPROC=$SLURM_NPROCS
echo Numero de procesos $NPROC
date
#Ruta del script para ejecutar el modulo en standalone
#Cada modulo tiene su propio script. Consultar el manual o
#ejecutar el siguiente comando :
# ls /dragofs/sw/restricted/0.2/software/BIOVIA/MaterialsStudio22.1/etc/
#Este es un ejemplo para CASTEP
CASTEP=/dragofs/sw/restricted/0.2/software/BIOVIA/MaterialsStudio22.1/etc/CASTEP/bin/RunCASTEP.sh
BASENAME= <El patron usado para identificar el conjunto de ficheros de salida y entrada en este caso de CASTEP>
$CASTEP -np $NPROC $BASENAME
date
- Plantilla para un script en más de un nodo:
#!/bin/bash
#SBATCH -p special
#SBATCH -N <Numero de nodos. Mayor que 1>
#SBATCH -n <Numero total de cpus>
#SBATCH --ntasks-per-node=<Número de procesadores a usar en cada nodo.>
#SBATCH --job-name=<Nombre del trabajo, por ejemplo 01-Job>
#SBATCH --mem-per-cpu=<Cantidad de memoria asignada por cpu, por defecto 4g>
#SBATCH --constraint=MS
(...continuar con la plantilla de un nodo...)
La cantidad de recursos del slurm depende del tipo de trabajo. Soliciar un número de inadecuado puede afectar de forma negativa a la ejecución de su trabajo.
Solo se debe utilizar la cola GPU con los módulos Forcite y Mesocite
Estas plantillas y otros ejemplos están disponible en el servicio Git del CSIC: